|
前回ソフト導入編で紹介したソフトはダウンロードできたでしょうか?
もう一度必要なソフトをここであげておきます。
1,ISISDraw
2.MOLDA
3.TINKER(Minimize.exe , Mm3.prm)
4.Rasmol
さて、今回はそれらを使って分子モデルを作成してみましょう!
<注意> これからはWindowsを使っている方を主体で書かせていただきます。私自身がWindowsですので。MACの方ごめんなさい。
まず構造式を描いてみましょう。ISISDraw
やMOLDAは触ってみたでしょうか?これらは分子構造式描画ソフトでISISは2D、MOLDAは3Dぽい2Dで分子を描くことが出来ます。MOLDAでも簡単な分子はすぐに描けるのですが、巨大分子や複雑な分子を描くにはISISの方が便利ですね。それでは簡単な分子ですが、ISISを使って「舟型シクロヘキサン」を書いてみましょう。
|
1.ISISDrawを起動する。
2.上部にあるボタンの左から4つ目のシクロヘキサンのボタンを選択する。
3.図1のようなシクロヘキサンを描くことが出来たと思います。
4.左のバーの上から6こめのボタンで結合にアップ、ダウンをつけます。上向きボンドツールを選んでシクロヘキサンを舟形にしてみてください。(図2) |
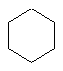

図1シクロヘキサン 図2舟形シクロヘキサン |
舟形シクロヘキサンは作れたでしょうか?それでは、次に今書いた分子をMOLDAで表示してみましょう。
|
1.ISISDrawで描いた分子を範囲選択する(一番上のボタンで分子を囲みます)
2.メニューの[ファイル]-[エクスポート]-[Molファイル]を選ぶ。
3.これを前回TINKERを保存したフォルダ[TINKER]へファイル名[boat.mol]で保存する(TINKERフォルダはMOLDAフォルダの中に作成してください)
4.MOLDAを開き、メニューの[ファイル]-[インポート]-[MDLmolFile]を選択し、先ほどエクスポートしたファイルboat.molをインポートする。
5.図3見たいな感じに表示されたら成功
6.マウスの右ボタンをドラッグして回転してみてください。 |
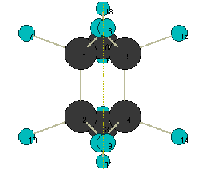
図3MOLDAで表示された舟形シクロヘキサン |
ここまで来たらあともうちょいです。とりあえず舟形の3Dシクロヘキサンが出来ました。でも、ちょっとおかしいですね。水素同士が近すぎるし、安定とは思えません。そこで、次はTINKERを使ってこれを最安定配座にしてみましょう。
|
1.MOLDAのメニューで[ファイル]-[エクスポート]-[TINKER]を選択する。
2.[Chenge Atom
type]というダイアログボックスが開いたら[OK]にする。
3.[保存]ダイアログボックスが開くので、そこで[boat.xyz]で保存する。(TINKERフォルダの中)
4.TINKERのMinimizeを起動する。(MS=DOSプロンプトが開く)
5.以下のように入力すると最適化計算が始まる。
Enter Cartesian Coordinate File Name
: boat.xyz
Enter Potential Parameter File Name
: mm3.prm
Enter RMS Gradient per Atom
Criterion[0.01]:0.01
6.終わると[TINKER]フォルダに[boat.xyz_2]というファイルが作成されます。 |
ここで、先ほどと同じようにMOLDAにインポートして表示してみます。
|
1.MOLDAメニューバーの[ファイル]-[インポート]-[TINKER]を選ぶ。
2.先ほど作成したboat.xyz_2というファイルを選択する。
3、図4のようなモデルが表示されたら成功。上の図2との違いわかるでしょうか?出来上がったら前と同じようにマウスの右ドラッグで分子を回してみてください。 |
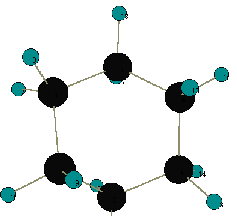
図4 MOLDAで表示した最適化した舟形シクロヘキサン |
MOLDAは3Dぽい2D?なので本格的な3Dグラフィック表示が出来るRasmolで表示してみましょう。
|
1.MOLDAメニューバーの[ファイル]-[エクスポート]-[Protein
Data Bank(.pdb)]を選ぶ。
2.[boat.pdb]で保存する。
3.Rasmolを起動する。
4.Rasmolメーニュバーの[File]-[open]を選ぶ。
5.先ほど保存した[boat.pdb]を選択する。
6.図5のようなモデルが表示されたら成功!左ドラッグで動かしてみたり、メニューバーのDisplayで分子表示を変えてみたりしてみてください。
|

図5 Rasmolで表示した舟形シクロヘキサン(この図はメニューバーの[Display]-[sticks]表示です。またWeb上で背景を変えています) |
うまくできたでしょうか?これらのフリーソフトを使っていろいろな分子を作成・表示できます。また、ここで使ったソフト以外にも、多数のフリーソフトやシェアソフト、市販ソフトがありそれらは互いに相関関係があります。
図6に簡単に表してみました。(他にもあります。これは一例です。)
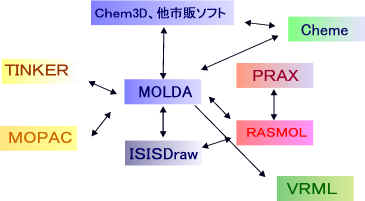
図6 化学ソフトの相関関係
黄:力場計算ソフト系 青:構造式描画ソフト系
赤:グラフィックソフト系(主にオフライン) 緑:グラフィックソフト系(主にオンライン)
今回分子モデルをつくるということだけに焦点をしぼって書いてみましたが、その背景もとても重要なことです。例えば、TINKERで構造最適化したプログラムはどのように最適化しているのか?等のことです。これらは分子動力学計算をコンピューターにやらせているだけです。mm2やmm3,EHMO,CINIDO,GAUSSIAN・・・とたくさんの計算方法、プログラムがあります。これらについては後々トピックス「分子動力学法」として紹介したいと思います。
有機って面白いよね!!
(2000/11 byブレビコミン)
|
